您将做什么¶
使用 NumPy 的 masked arrays 模块来分析 COVID-19 数据并处理缺失值。
您将学到什么¶
您将了解什么是掩码数组以及如何创建它们
您将看到如何访问和修改掩码数组的数据
您将能够决定在某些应用程序中使用掩码数组是否合适
您将需要什么¶
对 Python 有基本了解。如果您想复习一下,请查看 Python 教程。
对 NumPy 有基本了解
要在您的计算机上运行绘图,您需要 matplotlib。
什么是掩码数组?¶
考虑以下问题。您有一个包含缺失或无效条目的数据集。如果您要对此数据进行任何类型的处理,并且想跳过或标记这些不需要的条目而不只是删除它们,您可能需要使用条件语句或以某种方式过滤数据。 numpy.ma 模块提供了与 NumPy ndarrays 相同的功能,并增加了结构以确保无效条目不被用于计算。
来自 参考指南
掩码数组是标准 numpy.ndarray 和一个 **mask** 的组合。mask 要么是
nomask,表示关联数组中的所有值都是有效的;要么是一个布尔数组,它为关联数组中的每个元素确定该值是否有效。当 mask 的一个元素是False时,关联数组中相应的元素是有效的,并称为未被掩码。当 mask 的一个元素是True时,关联数组中相应的元素称为被掩码(无效)。
我们可以将 MaskedArray 视为以下内容的组合:
数据,一个任何形状或数据类型的常规
numpy.ndarray;一个与数据形状相同的布尔掩码;
一个
fill_value,一个可用于替换无效条目以返回标准numpy.ndarray的值。
它们何时有用?¶
在一些情况下,掩码数组比简单地消除数组中的无效条目更有用
当您想保留被掩码的值以供以后处理,而无需复制数组时;
当您必须处理多个数组,每个数组都有自己的掩码时。如果掩码是数组的一部分,您可以避免错误,代码也可能更紧凑;
当您有不同的缺失或无效值标志,并希望保留这些标志而不替换原始数据集中的值,但又不希望它们参与计算时;
如果您无法避免或消除缺失值,但又不想在您的操作中处理 NaN (Not a Number) 值时。
掩码数组也是个好主意,因为 numpy.ma 模块还附带了大多数 NumPy 统一函数 (ufuncs) 的特定实现,这意味着您仍然可以对掩码数据应用快速向量化函数和操作。输出将是一个掩码数组。我们将在下面看到一些实际操作的例子。
使用掩码数组查看 COVID-19 数据¶
从 Kaggle 可以下载一个包含 2020 年初 COVID-19 爆发的初始数据的数据集。我们将查看此数据的一个小子集,包含在文件 who_covid_19_sit_rep_time_series.csv 中。(注意,此文件已于 2020 年底被一个没有缺失数据的版本替换)。
import numpy as np
import os
# The os.getcwd() function returns the current folder; you can change
# the filepath variable to point to the folder where you saved the .csv file
filepath = os.getcwd()
filename = os.path.join(filepath, "who_covid_19_sit_rep_time_series.csv")数据文件包含不同类型的数据,组织方式如下:
第一行是标题行,(大部分)描述了下方各行中数据的含义,从第四列开始,标题是观察日期。
第二行到第七行包含汇总数据,与我们要检查的数据类型不同,因此我们需要将其排除在我们将要使用的数据之外。
我们要处理的数值数据从第 4 列、第 8 行开始,一直延伸到最右边的列和最下面的行。
让我们探索该文件中的数据,了解前 14 天的记录。为了从 .csv 文件收集数据,我们将使用 numpy.genfromtxt 函数,确保我们只选择包含实际数字的列,而不是包含位置数据的最初四列。我们还跳过了该文件的前 6 行,因为它们包含我们不感兴趣的其他数据。另外,我们将提取此数据的日期和位置信息。
# Note we are using skip_header and usecols to read only portions of the
# data file into each variable.
# Read just the dates for columns 4-18 from the first row
dates = np.genfromtxt(
filename,
dtype=np.str_,
delimiter=",",
max_rows=1,
usecols=range(4, 18),
encoding="utf-8-sig",
)
# Read the names of the geographic locations from the first two
# columns, skipping the first six rows
locations = np.genfromtxt(
filename,
dtype=np.str_,
delimiter=",",
skip_header=6,
usecols=(0, 1),
encoding="utf-8-sig",
)
# Read the numeric data from just the first 14 days
nbcases = np.genfromtxt(
filename,
dtype=np.int_,
delimiter=",",
skip_header=6,
usecols=range(4, 18),
encoding="utf-8-sig",
)在 numpy.genfromtxt 函数调用中,我们为数据的每个子集(整数 - numpy.int_ - 或字符字符串 - numpy.str_)选择了 numpy.dtype。我们还使用了 encoding 参数为文件选择了 utf-8-sig 编码(在 官方 Python 文档 中阅读有关编码的更多信息)。您可以从 参考文档 或 基本 IO 教程 中了解更多关于 numpy.genfromtxt 函数的信息。
探索数据¶
首先,我们可以绘制我们拥有的全部数据,看看它的样子。为了获得可读性强的图表,我们只选择一些日期在我们的 x 轴刻度 上显示。还请注意,在我们的绘图命令中,我们使用了 nbcases.T (nbcases 数组的转置),因为这意味着我们将文件中的每一行绘制为一条单独的线。我们选择绘制虚线(使用 '--' 线型)。有关更多信息,请参阅 matplotlib 文档。
import matplotlib.pyplot as plt
selected_dates = [0, 3, 11, 13]
plt.plot(dates, nbcases.T, "--")
plt.xticks(selected_dates, dates[selected_dates])
plt.title("COVID-19 cumulative cases from Jan 21 to Feb 3 2020")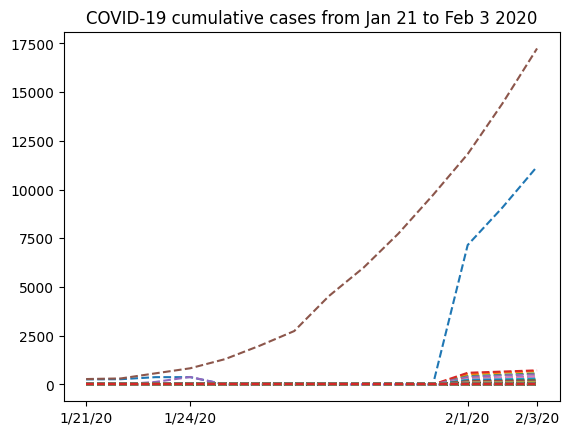
从 1 月 24 日到 2 月 1 日,图表显示出奇怪的形状。了解这些数据来自哪里会很有趣。如果我们查看从 .csv 文件中提取的 locations 数组,我们可以看到我们有两列,第一列包含地区,第二列包含国家名称。然而,只有前几行包含中国省份名称的数据。在此之后,我们只有国家名称。因此,将中国的所有数据分组到一个单独的行中是有意义的。为此,我们将从 nbcases 数组中仅选择 locations 数组的第二项对应中国(China)的行。接下来,我们将使用 numpy.sum 函数对所有选定的行求和(axis=0)。另外请注意,第 35 行对应于该国每天的总计数。由于我们想从省份数据中自己计算总和,所以我们必须先从 locations 和 nbcases 中删除该行。
totals_row = 35
locations = np.delete(locations, (totals_row), axis=0)
nbcases = np.delete(nbcases, (totals_row), axis=0)
china_total = nbcases[locations[:, 1] == "China"].sum(axis=0)
china_totalarray([ 247, 288, 556, 817, -22, -22, -15, -10, -9, -7, -4, 11820, 14410, 17237])这个数据有问题——我们在累积数据集中不应该有负值。怎么回事?
缺失数据¶
查看数据,我们发现:有一段时间 **缺失数据**
nbcasesarray([[ 258, 270, 375, ..., 7153, 9074, 11177], [ 14, 17, 26, ..., 520, 604, 683], [ -1, 1, 1, ..., 422, 493, 566], ..., [ -1, -1, -1, ..., -1, -1, -1], [ -1, -1, -1, ..., -1, -1, -1], [ -1, -1, -1, ..., -1, -1, -1]], shape=(263, 14))我们看到的所有 -1 值都来自 numpy.genfromtxt 尝试从原始 .csv 文件读取缺失数据。显然,我们不希望将缺失数据计算为 -1 ——我们只想跳过这个值,这样它就不会干扰我们的分析。导入 numpy.ma 模块后,我们将创建一个新数组,这次掩码无效值。
from numpy import ma
nbcases_ma = ma.masked_values(nbcases, -1)如果我们查看 nbcases_ma 掩码数组,这就是我们所拥有的
nbcases_mamasked_array( data=[[258, 270, 375, ..., 7153, 9074, 11177], [14, 17, 26, ..., 520, 604, 683], [--, 1, 1, ..., 422, 493, 566], ..., [--, --, --, ..., --, --, --], [--, --, --, ..., --, --, --], [--, --, --, ..., --, --, --]], mask=[[False, False, False, ..., False, False, False], [False, False, False, ..., False, False, False], [ True, False, False, ..., False, False, False], ..., [ True, True, True, ..., True, True, True], [ True, True, True, ..., True, True, True], [ True, True, True, ..., True, True, True]], fill_value=-1)我们可以看到这是一种不同的数组。如引言中所述,它具有三个属性(data、mask 和 fill_value)。请记住,mask 属性的值为 True 的元素对应于**无效**数据(在 data 属性中用两个破折号表示)。
让我们尝试查看排除第一行(中国湖北省的数据)后的数据,以便更仔细地查看缺失数据。
plt.plot(dates, nbcases_ma[1:].T, "--")
plt.xticks(selected_dates, dates[selected_dates])
plt.title("COVID-19 cumulative cases from Jan 21 to Feb 3 2020")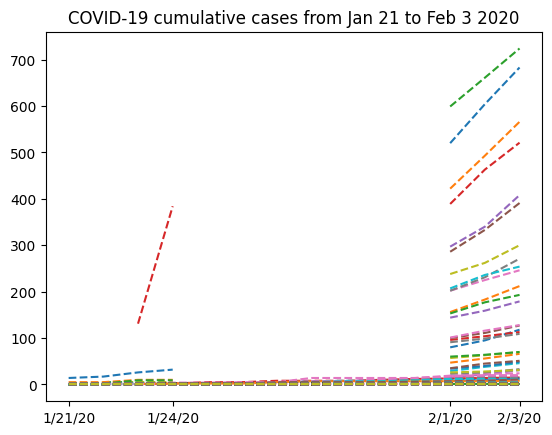
现在我们的数据已被掩码,让我们尝试对中国的所有病例进行求和。
china_masked = nbcases_ma[locations[:, 1] == "China"].sum(axis=0)
china_maskedmasked_array(data=[278, 309, 574, 835, 10, 10, 17, 22, 23, 25, 28, 11821, 14411, 17238], mask=[False, False, False, False, False, False, False, False, False, False, False, False, False, False], fill_value=999999)请注意,china_masked 是一个掩码数组,因此它具有与常规 NumPy 数组不同的数据结构。现在,我们可以直接通过 .data 属性访问其数据。
china_total = china_masked.data
china_totalarray([ 278, 309, 574, 835, 10, 10, 17, 22, 23, 25, 28, 11821, 14411, 17238])这样好多了:不再有负值。但是,我们仍然可以看到,在某些日期,累积病例数似乎在下降(例如从 835 到 10),这与“累积数据”的定义不符。如果我们更仔细地查看数据,我们可以看到,在中国大陆缺失数据的时期,香港、台湾、澳门和中国“未指定”地区的有效数据。也许我们可以从中国的总病例数中删除这些,以便更好地理解数据。
首先,我们将确定中国大陆地区在列表中的索引。
china_mask = (
(locations[:, 1] == "China")
& (locations[:, 0] != "Hong Kong")
& (locations[:, 0] != "Taiwan")
& (locations[:, 0] != "Macau")
& (locations[:, 0] != "Unspecified*")
)现在,china_mask 是一个布尔值数组(True 或 False);我们可以使用掩码数组的 ma.nonzero 方法来检查我们想要的索引是否正确。
china_mask.nonzero()(array([ 0, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 25, 26, 27, 28, 29, 31, 33]),)现在我们可以正确地对中国大陆的条目求和了。
china_total = nbcases_ma[china_mask].sum(axis=0)
china_totalmasked_array(data=[278, 308, 440, 446, --, --, --, --, --, --, --, 11791, 14380, 17205], mask=[False, False, False, False, True, True, True, True, True, True, True, False, False, False], fill_value=999999)我们可以用这些信息替换数据,并绘制一张新图,重点关注中国大陆。
plt.plot(dates, china_total.T, "--")
plt.xticks(selected_dates, dates[selected_dates])
plt.title("COVID-19 cumulative cases from Jan 21 to Feb 3 2020 - Mainland China")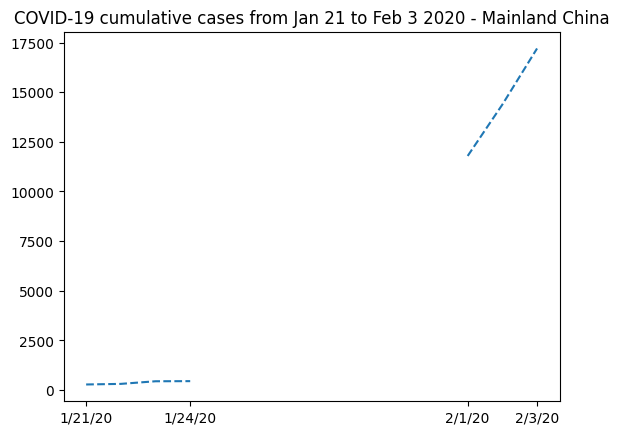
显然,掩码数组是这里的正确解决方案。如果没有误传曲线的演变,我们就无法表示缺失的数据。
拟合数据¶
我们可以想到的一个可能性是插值缺失数据来估算一月底的病例数。请注意,我们可以使用 .mask 属性来选择被掩码的元素。
china_total.mask
invalid = china_total[china_total.mask]
invalidmasked_array(data=[--, --, --, --, --, --, --], mask=[ True, True, True, True, True, True, True], fill_value=999999, dtype=int64)我们也可以通过对这个掩码使用逻辑非来访问有效条目。
valid = china_total[~china_total.mask]
validmasked_array(data=[278, 308, 440, 446, 11791, 14380, 17205], mask=[False, False, False, False, False, False, False], fill_value=999999)现在,如果我们想为这些数据创建一个非常简单的近似,我们应该考虑无效数据周围的有效条目。因此,我们首先选择数据有效的日期。请注意,我们可以使用 china_total 掩码数组的掩码来索引日期数组。
dates[~china_total.mask]array(['1/21/20', '1/22/20', '1/23/20', '1/24/20', '2/1/20', '2/2/20', '2/3/20'], dtype='<U7')最后,我们可以使用 `numpy.polynomial` 包的 拟合功能 来创建一个三次多项式模型,该模型尽可能好地拟合数据。
t = np.arange(len(china_total))
model = np.polynomial.Polynomial.fit(t[~china_total.mask], valid, deg=3)
plt.plot(t, china_total)
plt.plot(t, model(t), "--")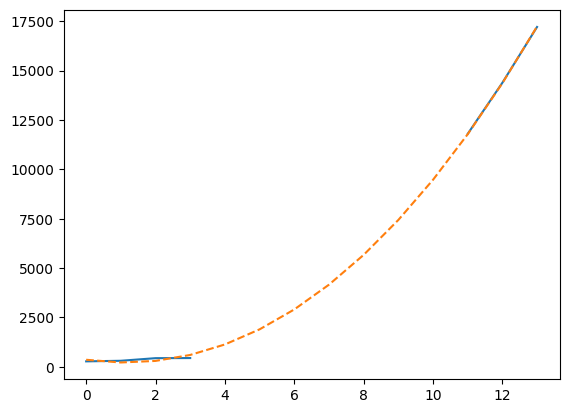
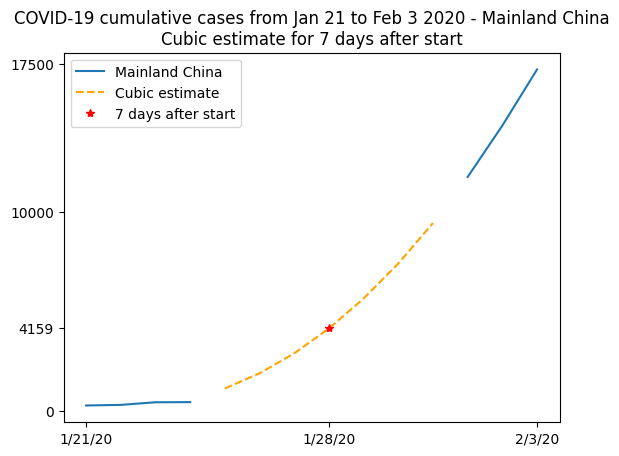
这张图不太容易读,因为线条似乎重叠在一起,所以我们将其在一个更详细的图中总结。当数据可用时,我们将绘制真实数据,并显示不可用数据的三次拟合,使用此拟合来计算 2020 年 1 月 28 日(即记录开始 7 天后)观察到的病例数的估计值。
plt.plot(t, china_total)
plt.plot(t[china_total.mask], model(t)[china_total.mask], "--", color="orange")
plt.plot(7, model(7), "r*")
plt.xticks([0, 7, 13], dates[[0, 7, 13]])
plt.yticks([0, model(7), 10000, 17500])
plt.legend(["Mainland China", "Cubic estimate", "7 days after start"])
plt.title(
"COVID-19 cumulative cases from Jan 21 to Feb 3 2020 - Mainland China\n"
"Cubic estimate for 7 days after start"
)
实践中¶
使用
numpy.genfromtxt添加-1到缺失数据不是问题;在这种特定情况下,用0替换缺失值可能也可以,但我们稍后会看到,这远非一个通用的解决方案。此外,可以通过使用usemask参数来调用numpy.genfromtxt函数。如果usemask=True,numpy.genfromtxt将自动返回一个掩码数组。
进一步阅读¶
本教程中未涵盖的主题可以在文档中找到。
参考¶
Ensheng Dong, Hongru Du, Lauren Gardner, An interactive web-based dashboard to track COVID-19 in real time, The Lancet Infectious Diseases, Volume 20, Issue 5, 2020, Pages 533-534, ISSN 1473-3099, Dong et al. (2020).
- Dong, E., Du, H., & Gardner, L. (2020). An interactive web-based dashboard to track COVID-19 in real time. The Lancet Infectious Diseases, 20(5), 533–534. 10.1016/s1473-3099(20)30120-1